|
解读纳米孔长读长测序分析和组装人类基因组的优势
2019-05-02 自1995年,流感嗜血杆菌首次成为首个进行全基因组测序的生物体以来,至今已获得了数以千计的完整基因组。然而,“完整基因组” 这一术语有些使用过度,因为多数测序基因组仍有许多测序“空白”。
据估计,人类尚有8%左右的基因组有待测序,这些 “空白”主要包括基因密度大、序列重复的异染色质DNA,这种DNA可见于着丝粒区和端粒区以及具有较大结构变异(SV)的区域。
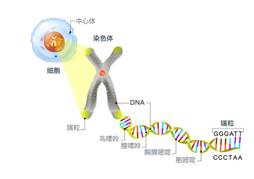
图1 据估计,约有8%的人类基因组还有待测序
建立真正完整的大基因组组装挑战极大,特别是考虑到重复区域在基因组的构成占比很大时。人体中约50%的基因组由重复序列组成,其他像开花植物等生物体DNA重复比例更高,占55%~83%。总体来说,要研究的基因组越大,全基因组测序和组装面临的考验也就更严峻。
纳米孔测序技术可处理所有待测序的DNA片段,对数以千计kb级大小的完整片段进行常规处理,这种长读长更有可能覆盖包含重复DNA和结构变异的整个区域。因此,纳米孔技术可提供更为完整的遗传变异图,同时实现单核苷酸变异(SNV)的单倍体分型、结构变异及重复区域的表征。
1 研究案例一:改善人类基因组组装
“我们已证实超长读长对组装连续性的有力影响,它能够解析那些已证实难以用短读长测序解析的基因组区域,包括端粒区、着丝粒和MHC等高度可变区。”
英国、美国和加拿大的研究者共同参与的多中心项目中,研究者选取了已充分表征的人类参考基因组NA12878,使用纳米孔测序,生成了91 Gb的数据,理论上可达30x人类基因组的覆盖度。
最终组装结果具有高度完整性和连续性,包含2886个contigs和一个NG50*约为3 Mb大小的contig。与当前NA12878参考组装相比,该团队识别出了899个结构性差异,这可能是先前未进行图谱表征的DNA的延伸。
*NG50值表示最长的连续序列,这种序列的长度至少大于单倍体基因组的一半。 † N50值表示基因组组装的连续性;其数值越高,基因组相连程度越高。
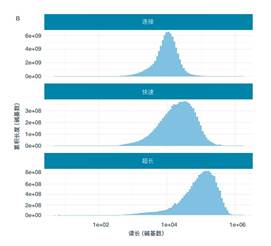
在研究高度重复的HLA I类区域(以难组装而出名)时,证实了纳米孔测序可实现连续性优良的基因组。不仅能所有HLA I类基因出现在一个大小为3 Mb的单超长读长测序证实了该方法可提高组装连续性,片段contig序列中,还可识别单倍体分型。相比较标准长读长实验方案(分别为10.6kb和3Mb),它交付了额外5x的基因组覆盖度,以及一个读长N50† 为99.7 kb 和NG50 为6.4 Mb的contig (图2)。获得的最长读长比对全长为882 kb。
图 2通过连接、快速及快速超长文库制备的读长产量。使用修改的苯酚:氯仿DNA提取法,按照Oxford Nanopore快速测序试剂盒方案 (超长),从细胞(而非购买的DNA) 中直接获取的最长读长。图片源自Jain (2017年)。
与短读长技术不同,纳米孔测序还可检测胞嘧啶甲基化(5-甲基胞嘧啶),并且结果显示与之前使用其它分析技术获取和发布的数据具有高度一致性。
2 研究案例二:用于人类基因组分析的长读长应用
“纳米孔测序在一个已经充分表征的人类参考基因组的几个区域识别出了一个大型的结构变异,但只使用短读长测序时这些大型的结构变异并不明显。”
乌得勒支大学的Wigard Kloosterman博士证明了纳米孔测序平台可以准确确定染色体碎裂的单倍型分型,染色体碎裂是高度群集的染色体DNA重排,在癌症和先天性疾病样本中都出现过(图3)。除了之前由短读长测序检测到的40个染色体碎裂断点,纳米孔测序平台还以更高的精度识别出更多其它的断点。
图 3纳米孔长读长测序能够鉴定串联重复,而串联重复在使用传统的短读长测序技术时并不明显。图片由荷兰乌得勒支大学Wigard Kloosterman博士提供。
同样,来自Genomics plc的Michael Simpson教授和维康信托基金会人类遗传中心(WTCHC)的研究者们也利用纳米孔测序研究了一个样本中的单倍型分型,该样本显示出与血细胞减少和小脑性共济失调相关的免疫功能紊乱。维康信托基金会人类遗传中心的团队成功证明了两个之前表征的SAMD9L基因中相距2.2kb的新生错义变异实际位于同一单倍型上。
维康信托基金会人类遗传中心和Genomicsplc的团队还使用纳米孔测序在一个已经充分表征的人类基因组NA12878的几个区域上识别出了大型的结构变异,而仅使用短读长测序时,这些大型的结构变异并不明显——这进一步彰显出了长读长测序技术的优势(图4)。
图 4通过纳米孔测序产生的长读长能够在已经充分表征的人类参考基因组NA12878的10号染色体上识别出一个7kb的大缺失,这一缺失在使用短读长测序时并不明显。数据由Genomicsplc和维康信托基金会人类遗传中心的Michael Simpson提供
您也可以扫描下方二维码下载纳米孔应用综述白皮书,了解更多纳米孔测序的研究案例。
Oxford Nanopore的产品现已接受网上订购,请打开“阅读原文“或浏览以下网址在线订购:htt p://store.nanoporetech.com/cn 。
参考文献
[1] Fleischmann,R.D.等人(1995),《流感嗜血杆菌全基因组随机测序和组装》,《科学》269(5223): 496-512 [2] Chaisson, M.J.P.等人(2015),《人类基因组基因变异和全新组装》,《遗传学自然评论》16(11):62740 [3] Treangen, T.J.和Salzberg, S.L.(2011)《重复DNA与下代测序:计算挑战和解决方案》《遗传学自然评论》13(1):36-46.doi: 10.1038/nrg3117 [4] Macas, J.等人(2015)《23个植物基因组重复DNA的深入研究披露了野碗豆族基因组大小变异的来 源》《公共科学图书馆期刊》 10(11): e0143424.doi:10.1371/journal.pone.0143424 [5] Loman, N.(2017) 950kb,yeah! [推特]3月8日网址:https://twitter.com/pathogenomenick/status/839604187100934144[访问日期:2017年7月20] [6] Jain, M.等人(2017)《人类基因组超长读长纳米孔测序和组装》bioRxiv doi.org/10.1101/128835 [7] Chaisson, M.J.P. 等人 (2015) 《人类基因组的遗传变异与从头组装》,《遗传学自然评论》16(11):627–40 [8] Simpson, M. (2016) 《利用纳米孔技术对人类基因组进行测序》,介绍。网址:https:// nanoporetech.com/events/ncm16#193976224 [访问日期:2017年1月5日]
制版人:半夏
精选留言 暂无... |