|
两篇PNAS报道植物RNA新修饰:NAD+-capped RNA原创: 虞莎 BioArt植物
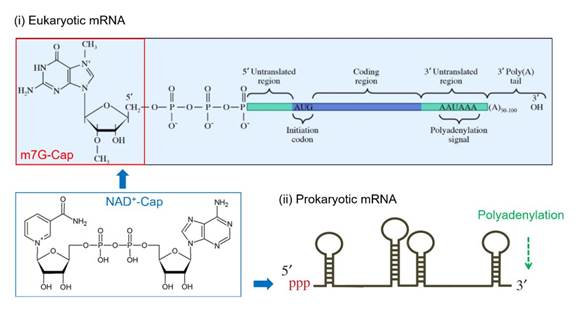
2019-00-27 真核细胞的mRNA在转录 (co-transcriptional) 以及转录后 (post-transcriptional) 存在复杂而又精细的调控,包括5’加帽(m7G-cap)、RNA剪切(splicing)、3’加尾(polyadenylation)、RNA编辑和修饰。帽子结构 (m7G-cap) 作为所有真核生物mRNA的特征,它一方面保护mRNA不被核酸外切酶降解,同时也作为一个重要的“桥梁分子”,招募与之相互作用的蛋白 (cap-binding protein), 调控mRNA的加工、运输以及依赖于m7G-cap的蛋白翻译过程【1】。与真核生物不同,原核细胞mRNA的转录和翻译过程高度偶联,一般认为原核生物的mRNA在转录后很少发生加工,其结构与真核mRNA存在很大的差异 (图1)。
图1. 真核和原核生物mRNA的结构以及NAD+-cap
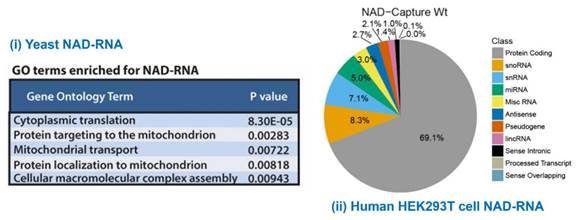
然而,自1992年大肠杆菌的Poly(A)聚合酶被克隆之后,原核细胞的mRNA也被证实存在由多聚腺苷酸构成的poly(A)尾巴,负责调控mRNA的稳定性【2】。近几年的研究还发现,原核生物mRNA的5’端并不只是焦磷酸基团,一种三磷酸腺苷(ATP)的衍生物——烟酰胺腺嘌呤二核苷酸 (Nicotinamide adenine dinucleotide, NAD) 构成了原核mRNA的5’帽子结构(NAD+-cap, 图1) 【3】。由于NAD与ATP结构相似,它作为非典型的核苷酸能够被细菌的RNA聚合酶识别,并且从头起始转录过程 (de novo transcription initiation)【4】。更有意思的是,NAD+-cap不仅存在于细菌中,在酵母和人类细胞中也发现了NAD+-capped RNA (以下简称为NAD-RNA),这些RNA主要由核基因和线粒体基因转录而来,并且大部分是蛋白质编码基因的产物 (图2) 【5,6】。
图2. 酵母和人类细胞中NAD+-capped RNA的组成 (参考 【5,6】)
作为RNA 5’帽子结构的新成员,NAD+-cap的功能目前尚不清楚。大肠杆菌中的研究结果表明,与真核生物的m7G-cap类似,NAD+-cap能够抵抗RNA焦磷酸水解酶 (RNA pyrophosphohydrolase, RppH) 对mRNA 5’焦磷酸的水解,增强mRNA的稳定性【7】。在细菌和HEK293T细胞中,都存在一类识别NAD+-cap的酶 (图3),它们能够将NAD+-cap从mRNA的5’末端移除,降低mRNA的稳定性【6,7】。这一过程被称之为“deNADding” (借鉴了真核细胞中mRNA的decapping)。NAD+-cap在原核和真核生物中的保守存在以及动态的调节机制,提示我们这一新型的帽子结构在细胞中可能具有重要的作用,与m7G-cap一样,NAD+-cap对于mRNA (尤其是原核细胞mRNA) 的功能或许至关重要。
图3. NudC和DXO分别在原核和真核细胞中移除mRNA的NAD+-cap (参考 【6,7】)
2019年5月29日,PNAS杂志发表了两篇拟南芥中NAD+-capped RNA的研究工作 【8,9】。这两篇论文由加州大学河滨分校(University of California, Riverside) 陈雪梅教授实验室以及香港浸会大学 (Hong Kong Baptist University) 夏亦荠教授实验室完成。继NAD-RNA在细菌、酵母和人类细胞中被报道之后,植物中也发现了NAD-RNA,为NAD+-cap在进化过程中的保守性和重要作用提供了又一项有力的证据。
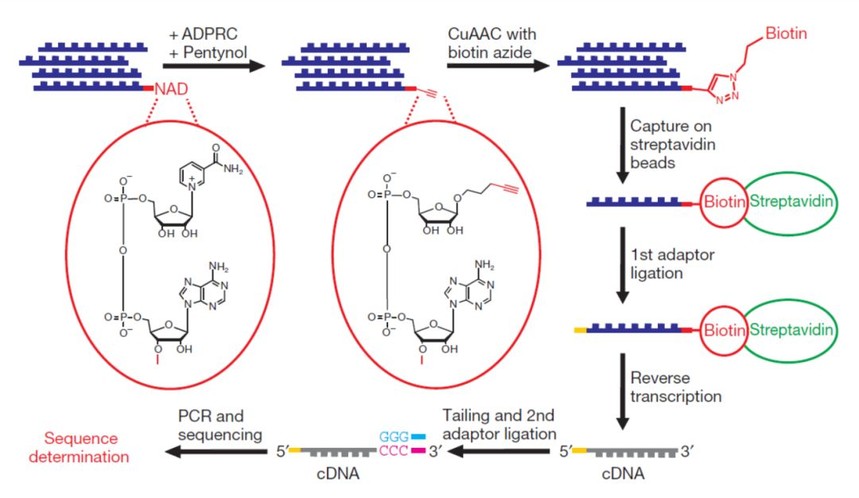
植物细胞中NAD+-cap的鉴定 研究人员首先证实NAD+-cap在植物细胞中是存在的,由于该帽子结构位于mRNA的5’最末端,他们利用核酸酶 P1将拟南芥的RNA水解为单个核苷酸,通过液相色谱-质谱联用 (liquid chromatography-mass spectrometry, LC/MS)鉴定到了NAD分子。接下来,他们利用高通量测序,在植物细胞的转录组中研究NAD-RNA的种类、丰度及其参与的生物学过程。高通量测序鉴定NAD-RNA的方法最早在细菌中报道,包括两步重要的化学反应: (1) 利用ADP-核糖环化酶 (Adenosine diphosphate-ribosylcyclase, ADPRC) 催化NAD和炔基化合物 (alkynyl alcohols) 发生转糖苷作用(transglycosylation),将NAD末端的氨基替换成炔基(alkynes);(2) 含有炔基的NAD进一步和生物素叠氮化物(biotin azides)发生“点击化学(Click chemistry)”的叠氮炔环加成反应(Copper-catalyzed Azide-Alkyne Cycloaddition, CuAAC),将生物素特异的标记在NAD-RNA上。最后通过“生物素—链霉亲和素”系统的纯化和富集,构建专一的NAD-RNA文库并进行高通量测序,这一方法称为“NAD captureSeq”(图4)【7】。
图4. NAD captureSeq的反应机理和建库过程 (参考【7】)
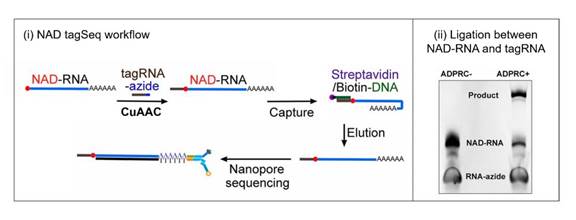
NAD captureSeq利用点击化学特异地标记、分离和富集NAD-RNA,然而在建库过程中RNA与链霉亲和素的非特异结合会对测序结果造成干扰,产生假阳性。同时,点击化学反应也会导致RNA链的断裂,使得部分RNA的信息在建库过程中丢失。基于以上不足,夏亦荠教授团队对NAD captureSeq的方法进行了改进,他们在第二步的反应中,将生物素叠氮化物(biotin azides)替换成一段3’末端含有叠氮基的单链RNA(tagRNA)。通过叠氮炔环加成反应(CuAAC),tagRNA与NAD-RNA共价连接形成一个嵌合分子,再利用与tagRNA序列互补的探针对NAD-RNA进行富集。随后,夏亦荠教授团队采用了第三代单分子测序技术“纳米孔测序(nanopore sequencing)”。该方法直接对RNA分子进行“从头到尾”的全长测序,不仅能够检测NAD-RNA的种类和丰度,通过tagRNA的序列还能进一步确定NAD+-cap修饰的位点。他们将该方法命名为“NAD tagSeq” (图5)。
图5. NAD tagSeq建库过程(i)以及tagRNA和NAD-RNA的连接(ii) (参考 【8】)
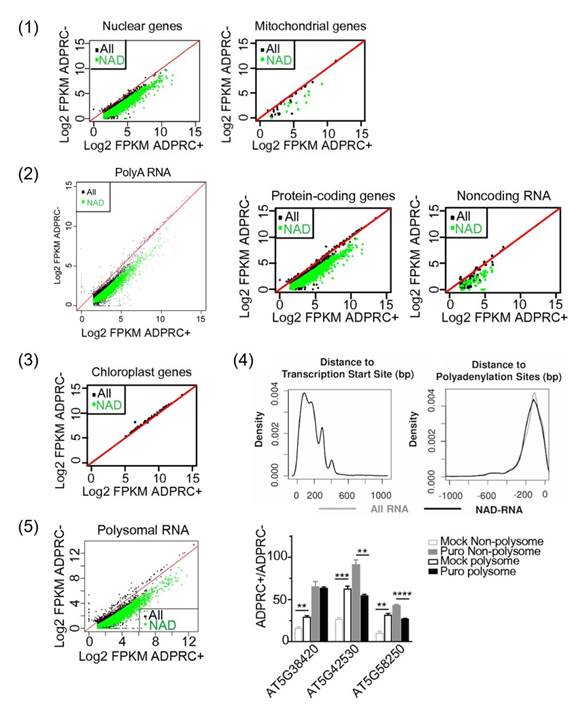
植物细胞中NAD-RNA的构成 两篇文章分别采用了NAD captureSeq和NAD tagSeq,对拟南芥幼苗和花序中的NAD-RNA进行了研究。共有1,712种NAD-RNA被两个实验室同时鉴定到,说明NAD+-cap修饰在植物细胞中是广泛存在的。综合两篇文章对测序结果的分析,我们发现植物细胞中的NAD-RNA具有以下特征: (1) 大部分是细胞核基因的转录产物,也有小部分由线粒体基因组编码,这与酵母以及人类细胞中NAD-RNA的构成类似; (2) 植物NAD-RNA含有poly(A)尾巴,主要由蛋白质编码基因转录而来,也有少量的非编码基因的产物;(3) 植物细胞的叶绿体基因组并不编码NAD-RNA,叶绿体和线粒体中NAD+/NADH的不同可能造成了NAD-RNA在这两个细胞器中的差异表达;(4) 与m7G 相比,NAD+-cap大多位于转录起始位点下游30-400个核苷酸的位置,即NAD-RNA的5’ UTR比m7G-capped RNA更短,而二者mRNA 3’末端的多聚腺苷酸位点却没有差异; (5) NAD-RNA存在于植物细胞的多聚核糖体组分(polysome fraction)中,利用嘌呤霉素(puromycin)阻断蛋白质的翻译会减少NAD-RNA在多聚核糖体中的富集,说明NAD-RNA可能参与了蛋白质的翻译过程 (图6)。
图6. 植物NDA-RNA的特征以及潜在的生物学功能 (参考 【8,9】)
此外,通过对植物的NAD-RNA进行GO功能注释,研究人员发现NAD-RNA富集于氧化应激 (oxidative stress)、光合作用、氧化还原稳态(redox homeostasis) 等生物学过程,提示我们,NAD-RNA可能调控了植物细胞对外界环境的感应,以及细胞内的氧化还原状态和能量代谢过程。关于NAD-RNA的产生机制、稳定性以及生物学功能等还有待进一步的探索和研究。
参考文献: [1] A. Ramanathan, G. B. Robb, S. H. Chan, mRNA capping: Biological functions and applications. Nucleic Acids Research. (2016). [2] E. Hajnsdorf, V. R. Kaberdin, RNA polyadenylation and its consequences in prokaryotes. Philosophical Transactions of the Royal Society B. (2018). [3] Y. G. Chen, W. E. Kowtoniuk, I. Agarwal, Y. Shen, D. R. Liu, LC/MS analysis of cellular RNA reveals NAD-linked RNA. Nature Chemical Biology. (2009). [4] J. G. Bird et al., The mechanism of RNA 5’ capping with NAD+, NADH and desphospho-CoA. Nature. (2016). [5] R. W. Walters et al., Identification of NAD+-capped mRNAs in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. (2017). [6] X. Jiao et al., 5’ End Nicotinamide Adenine Dinucleotide Cap in Human Cells Promotes RNA Decay through DXO-Mediated deNADding. Cell. (2017). [7] H. Cahová, M. L. Winz, K. Höfer, G. Nübel, A. Jäschke, NAD captureSeq indicates NAD as a bacterial cap for a subset of regulatory RNAs. Nature. 519, 374–377 (2015). [8] Y. Wang et al., NAD+-capped RNAs are widespread in the Arabidopsis transcriptome and can probably be translated. Proceedings of the National Academy of Sciences. (2019). [9] H. Zhang et al., NAD tagSeq reveals that NAD+-capped RNAs are mostly produced from a large number of protein-coding genes in Arabidopsis. Proceedings of the National Academy of Sciences. (2019).
精选留言 暂无... |